一种提高植物再生、转化效率的新策略
来源:《植物生物技术杂志》
作者:Jianying Li等
时间:2018-07-26
华中农业大学张献龙教授领衔的棉花团队发表了题为“Multi-omics analyses reveal epigenomics basis for cotton somatic embryogenesis through successive regeneration acclimation (SRA) process”的文章,开发了一种能提高棉花再生、转化效率的新策略,并对这一策略的表观遗传学机制进行了探讨,该研究结果丰富了对植物再生过程中的DNA甲基化分子机制的认识,为提高植株再生效率提供可行的策略,对于改善低再生、转化效率的植物物种的遗传转化具有借鉴意义。
这一研究成果公布在7月12日的Plant Biotechnology Journal杂志上,这项研究开发了一种通过连续组织培养、再生驯化策略提高棉花再生、转化效率的新策略,并利用多重组学研究手段解析了这一现象的的表观遗传学基础,李健英为论文第一作者,金双侠教授为论文通讯作者。
许多转基因作物是通过农杆菌介导的遗传转化、植株再生(器官发生,体细胞胚胎发生等)方式产生,这个过程非常耗时且高度依赖于基因型。以棉花(Gossypium spp.)遗传转化为例,目前只有少数几个材料能够通过组织培养获得再生植株,而且整个转化、植株再生过程需要1年甚至1年半的时间。再生效率低、转化周期长,已经成为制约棉花功能基因组研究的主要障碍之一。
目前国际上只有 Corker201/310/312,泗棉3号, 中12等少数棉花材料能够通过体细胞胚胎发生途径获得再生植株,但这些材料的农艺性状都普遍较差,且再生转化效率偏低,转化周期长。
这一研究团队自从上个世纪90年开始,一直致力于棉花遗传转化体系的优化和改良。先后对数百个陆地棉材料进行了再生能力的比较,筛选到一个优良的受体材料YZ-1 (金双侠 , 张献龙等 2006,Identification of a novel elite genotype for in vitro culture and genetic transformation of cotton )。YZ-1作为受体材料的转化效率、再生周期较以往的棉花受体材料都有较大提升,其转化效率提高到25%以上,是国际上棉花遗传转化模式材料-柯字棉系列(Corker201/310/312)转化效率(3-5%)的5倍左右,再生周期由18个月缩短到8-10个月左右,YZ-1目前已经成为国际上最重要的棉花转化受体之一。
但YZ-1存在生育期短,综合农艺性状差,不耐高温等不足,因此亟待培育新的、优良的棉花转化受体材料来代替YZ-1。
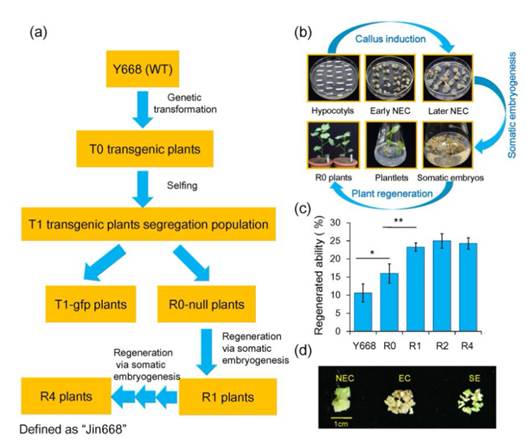
在这项研究通过对一个2000年以后选育的、农艺性状较好的棉花主栽品种为研究对象,经过连续再生驯化(SRA)提高棉花的转化效率策略(通过获取经过体细胞胚胎发生过程获得的再生植株的种子,作为下一次组织培养的受体材料,连续重复这一过程多次), 获得了高遗传转化效率的材料,研究人员把它命名为Jin668 (转化效率约20%-25%)远高于母体材料Y668(WT)(8-10%)(金双侠,张献龙等, 专利申请号:201510836230.6)。
在植物组织培养阶段,很多与植物再生相关的内源基因会经历DNA甲基化和组蛋白修饰等表观遗传因子的改变,为了研究SRA潜在表观遗传机制,我们构建了再生植株和野生型以及Jin668体细胞胚胎发生三个特定时期(NEC,EC, SE)全基因组上单碱基分辨率的DNA甲基化图谱。结果研究显示Jin668与R0相比,显示出低的CHH DNA甲基化,再生后代中整体甲基化呈下降趋势。
在体细胞胚胎发生阶段,NEC到EC过程中DNA甲基化呈现上升的趋势与RdDM途径和H3K9me2途径协同调控,EC到SE阶段DNA甲基化呈现下降,与RdDM显著相关, 不与H3K9me2相关, 进一步分析发现在EC到 SE过程中DNA去甲基化酶ROS1和DME显著上调,该过程可能高度依赖于DNA甲基化途径。
研究人员通过对差异表达基因和启动子区域的DMR分析发现,57个基因在启动子区域低甲基化可以激活生长素激素相关和WUSCHEL等标记基因表达。在NEC时期外施DNA甲基化抑制剂增加了胚胎数量,激活了WOX11、CKX7、KNOX2等基因表达,促进了体细胞胚胎的发生。因此,他们推测在SRA过程中低(去)甲基化有助于体细胞胚胎发生相关基因的激活,进而提高植物再生能力,可能是SRA策略起作用的重要原因之一。
Jin 668优良受体材料的鉴定将会为棉花功能基因研究提供新的重要的支撑材料。对SRA机制的研究丰富了我们对植物再生过程中DNA甲基化分子机制的认知,为提高植株再生效率提供可行的策略,对于改善低再生效率其他植物物种具有重要意义。(来源:生物通)
Multi-omics analyses reveal epigenomics basis for cotton somatic embryogenesis through successive regeneration acclimation (SRA) process
Abstract Plant regeneration via somatic embryogenesis is time‐consuming and highly genotype‐dependent. The plant somatic embryogenesis process provokes many epigenetics changes including DNA methylation and histone modification. Recently, an elite cotton Jin668, with an extremely high regeneration ability, was developed from its maternal inbred Y668 cultivar using a Successive Regeneration Acclimation (SRA) strategy. To reveal the underlying mechanism of SRA, we carried out a genome‐wide single‐base resolution methylation analysis for non‐embryogenic calluses (NECs), ECs, somatic embryos (SEs) during the somatic embryogenesis procedure and the leaves of regenerated offspring plants. Jin668 (R4) regenerated plants were CHH hypomethylated compared with the R0 regenerated plants of SRA process. The increase of CHH methylation from NEC to EC were demonstrated to be associated with the RNA‐dependent DNA methylation (RdDM) and the H3K9me2‐dependent pathway. Intriguingly, the hypomethylated CHH differentially methylated regions (DMRs) of promoter activated some hormone‐related and WUSCHEL‐related homeobox genes during the somatic embryogenesis process. Inhibiting DNA methylation using zebularine treatment in NEC increased the number of embryos. Our multi‐omics data provide new insights into the dynamics of DNA methylation during the plant tissue culture and regenerated offspring plants. This study also reveals that induced hypomethylation (SRA) may faciliate the higher plant regeneration ability and optimize maternal genetic cultivar.
原文链接:https://onlinelibrary.wiley.com/doi/epdf/10.1111/pbi.12988




