破解侵袭性乳腺癌的独特标签
来源:Scientific Reports
作者:Sagarika Banerjee等
时间:2015-10-28
从遗传学层面上来说,癌症是正常细胞功能出错的结果。这个事实为人所知已经有一段时间,但越来越多的证据表明,人类微生物组——每个人体内不同的微生物种群,可能在促成癌症、甚至直接引发一些形式癌症的过程中,发挥关键的作用。最近,来自宾夕法尼亚大学医学院的一个研究小组,在Erle S. Robertson博士和James C. Alwine博士的带领下,首次表明了两种微生物标签和三阴性乳腺癌(TNBC,乳腺癌最具侵袭性的形式)之间的一种关系。这一研究成果本周发表于《Scientific Reports》。
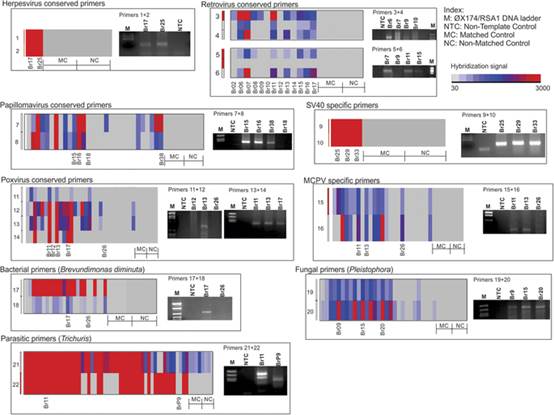
Abramson癌症中心核心服务副主任、癌症生物学教授Alwine说:“病毒和其他微生物,可能与癌症有更多的关系。”他们使用一种称为PathoChip的芯片技术(包含60000个分子探针),来识别所有已知的病毒和细菌、真菌、寄生虫和其他微生物。微生物学教授Robertson及其同事,对来自100 名TNBC患者的组织样本,进行了筛选。
他们还检测了40个匹配和不匹配的对照组(匹配的对照组是来自于TNBC患者的非肿瘤组织;不匹配的照组组来自于健康者的乳腺组织)。
该团队发现一个独特的微生物标签,可以将TNBC组织与正常样本区分开来,可进一步划分为两大类,一种主要是病毒性的,另一种主要是细菌性的,有一些真菌和寄生虫。
Robertson指出:“如果我们仔细观察,我们也可以在这些主要群组中发现一些更小的群集,可让我们对这些集群的独特标识符,有一定了解。我们发现,有大约30种生物,可提供一种特定类型的标签,为我们开发诊断工具提供线索。”
在检测到的病毒中,最常见的有疱疹病毒、副痘病毒、逆转录病毒、嗜肝病毒、多瘤病毒和乳头状瘤病毒。重要的细菌标签包括,化脓隐秘杆菌、短波单胞菌、鞘脂杆菌和芽孢杆菌,而真菌物种Pleistophora和Piedra、寄生生物Foncecaea和Trichuris是最明显的。
Alwine强调,在TNBC组织中检测到这些和其他病原体,并不一定意味着它们真正能够导致癌症。他指出:“有很多不同的方式来看待这一点。极有可能是,我们正在研究的一些生物体可能有致病作用,但我们不知道具体是哪种。在它被许多实验彻底地测试之前,我们不会知道。”一种可能是,生物体可能向细胞微环境中添加了一些东西,帮助受损细胞成为恶性或将它们推向癌症的边缘。另外,某些生物体可能仅仅发现,肿瘤组织是一个良好的环境,而根本没有直接参与癌症。
在这两种情况下,寻找一个与癌症相关的不同微生物标签,可提高新的诊断可能性的前景。Alwine解释说:“我们将这一标签视为在更早阶段诊断癌症的一种潜力。”
Robertson说:“我们正在研究不同的癌症,以查明特定的癌症是否有特定的标签。我们也在尝试用一种方法来研究血液样本,探究我们是否有可能开发一种测试法,来检测这些标签,这可能提供一个早期的监测系统,用于识别哪些患者最有可能患上这些恶性肿瘤。”
Alwine说:“微生物标签也可能有重要的治疗意义。这些微生物中有很多可以代谢药物,所以它们的存在会降低某些药物的效果,并可能指导我们确定使用什么方法来治疗癌症。”研究小组下一步计划探索这些有趣的前景。
Alwine说:“我们并不是说,所有这些生物都可直接导致癌症。我们正在试图确定特定的肿瘤标志,以便我们能想出办法来治疗、控制或更改癌症,或者治疗肿瘤相关的微生物,以在最初预防这些恶性肿瘤。我们现在开始更具体地研究,这些生物实际上在干什么。”(来源:生物通 王英)
Distinct microbiological signatures associated with triple negative breast cancer
Abstract Infectious agents are the third highest human cancer risk factor and may have a greater role in the origin and/or progression of cancers, and related pathogenesis. Thus, knowing the specific viruses and microbial agents associated with a cancer type may provide insights into cause, diagnosis and treatment. We utilized a pan-pathogen array technology to identify the microbial signatures associated with triple negative breast cancer (TNBC). This technology detects low copy number and fragmented genomes extracted from formalin-fixed paraffin embedded archival tissues. The results, validated by PCR and sequencing, define a microbial signature present in TNBC tissue which was underrepresented in normal tissue. Hierarchical clustering analysis displayed two broad microbial signatures, one prevalent in bacteria and parasites and one prevalent in viruses. These signatures demonstrate a new paradigm in our understanding of the link between microorganisms and cancer, as causative or commensal in the tumor microenvironment and provide new diagnostic potential.
原文链接:http://www.nature.com/articles/srep15162




