研究揭示双链DNA中错配碱基自发翻转规律
来源:PNAS
作者:赵新生等
时间:2014-05-30
北京大学生物动态光学成像中心赵新生、高毅勤两个课题组通力合作,近日在《美国科学院院刊》上发表重要论文,利用新颖的单分子实验手段重新测算了双链DNA错配碱基自发反转的速率,并运用动力学模拟方法对其分子机理进行了深入研究。对于阐明酶对碱基进行修复的分子机理具有重要价值。
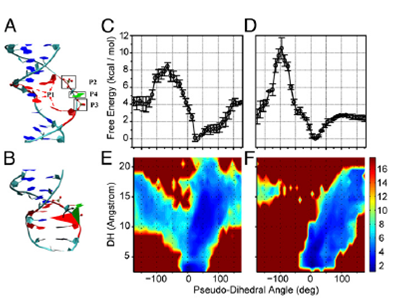
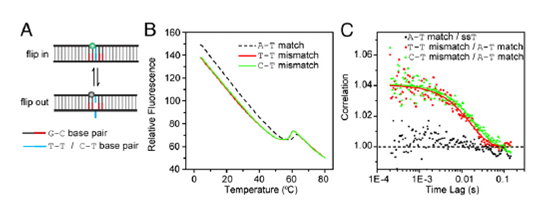
如果在双链DNA中有一错配的碱基对,其中的一个碱基是否可以自发地翻转出来?如果可以,其速率是多少?这不仅是DNA运动的一个基本问题,而且具有重要的生物学意义。细胞自身和环境的各种因素会造成DNA的损伤而导致错配碱基对的形成。碱基错配会引发细胞的病变甚至死亡,因此需要通过酶的作用及时给予修复。 为了回答酶如何找到错配位点进而完成修复这一问题,需要了解双链DNA中错配碱基自发翻转的动力学。过去仅有的实验数据来源于NMR基于质子交换速率的测量,但这一解释受到理论研究的强烈质疑。人们一直期待着用更可靠的实验方法对双链DNA中错配碱基自发翻转速率进行新的测量。
赵新生、高毅勤两个课题组与北大合成与功能生物分子中心的何川和伊成器两个课题组紧密合作,利用新颖的单分子实验手段得到错配碱基自发翻转速率,并用动力学模拟方法对其分子机理进行了深入研究。他们测得的速率比NMR声称的慢了4个数量级,原因是NMR所测得的速率并非碱基自发翻转速率,而是碱基在稳定构型附近摆动的速率。该成果有望大大提高酶对碱基进行修复的分子机理的理解。
赵新生、高毅勤教授同时担任北大化学学院长江特聘教授。尹延东和杨立江为该论文的共同第一作者。该工作得到了科技部973计划和国家自然科学基金项目的资助。(来源:科学网)
Dynamics of spontaneous flipping of a mismatched base in DNA duplex
Abstract DNA base flipping is a fundamental theme in DNA biophysics. The dynamics for a B-DNA base to spontaneously flip out of the double helix has significant implications in various DNA–protein interactions but are still poorly understood. The spontaneous base-flipping rate obtained previously via the imino proton exchange assay is most likely the rate of base wobbling instead of flipping. Using the diffusion-decelerated fluorescence correlation spectroscopy together with molecular dynamics simulations, we show that a base of a single mismatched base pair (T–G, T–T, or T–C) in a double-stranded DNA can spontaneously flip out of the DNA duplex. The extrahelical lifetimes are on the order of 10 ms, whereas the intrahelical lifetimes range from 0.3 to 20 s depending on the stability of the base pairs. These findings provide detailed understanding on the dynamics of DNA base flipping and lay down foundation to fully understand how exactly the repair proteins search and locate the target mismatched base among a vast excess of matched DNA bases.
原文链接:http://www.pnas.org/content/early/2014/05/14/1400667111.full.pdf+html




