成都生物所王斌团队整合转录组学与肽组学方法揭示无指盘臭蛙皮肤内源性分泌肽的超高多样性
来源:生物多样性保护中心
作者:刘炯宇
时间:2025-12-08
两栖动物皮肤富含内源性分泌肽(ESPs,Endogenous Secretory Proteins),在多种生理机能中发挥关键作用。然而,传统分离纯化与cDNA文库法难以全面获取内源性分泌肽,而常规无靶向转录组方法因信号肽预测假阴性而系统性遗漏大量候选序列,其根源在于起始密码子被误注释于5′非翻译区(5′-UTR),致使前体蛋白N端序列异常延长,从而引发信号肽预测失效。中国科学院成都生物研究所王斌团队创新开发了一种整合转录组学与肽组学的发现流程,有效纠正上述假阴性问题,将潜在内源性分泌蛋白鉴定效率提升61.6%。
该流程由三个部分组成(图1)。第一部分(绿色)主要是在构建转录组并获得预测蛋白集之后,调用自研脚本ORF_all.sh进行分析:首先构建过滤蛋白集(FPS,Filtered TransDecoder Protein Set);然后系统生成潜在的移码开放阅读框蛋白(ORF_shift proteins);再从中筛选预测为分泌蛋白的序列,形成优化分泌蛋白集(OSPS,Optimized Secretory Protein Set),随后合并FPS与OSPS并去除重复序列,最终得到用于搜库的蛋白数据库(SPS,Search Protein Set)。第二部分(蓝色)涉及蛋白水平的肽段鉴定,包括从分泌物中提取多肽、经液质联用并搜库鉴定,随后通过自编脚本Peptide.py对搜库结果精细化处理。该脚本可去除中低FDR置信度的蛋白和含奇数去氢半胱氨酸的肽段,整合肽段异构体并累积丰度,并在各主蛋白的特异性肽段中筛选丰度最高者作为候选成熟肽。第三部分(黄色)则通过整合蛋白和转录水平信息进行肽组发现,包括通过多步骤流程提取内源性分泌蛋白,完成信号肽分配与两步同源性BLASTP搜索,并最终结合所有分析结果进行人工注释。
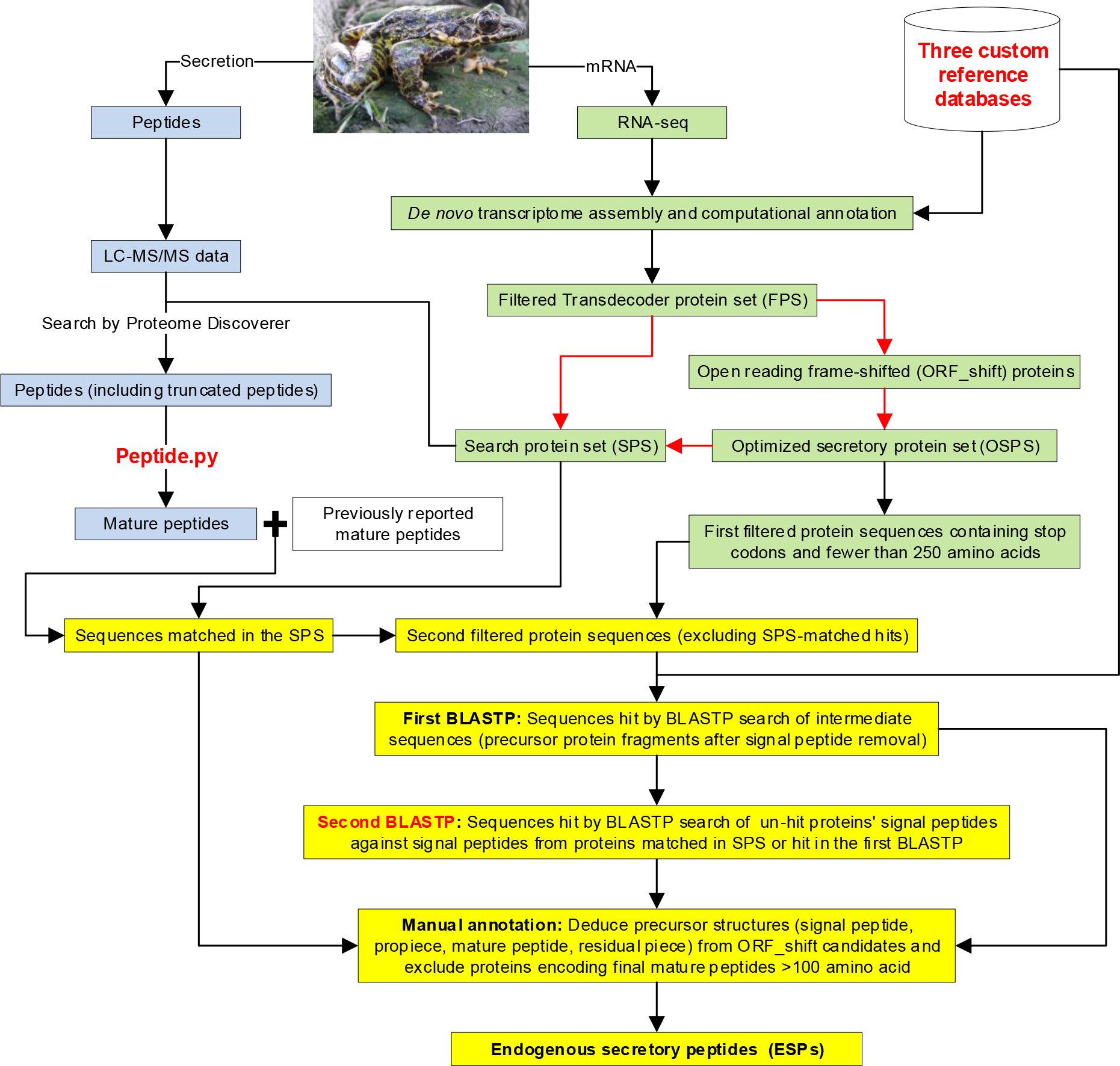
图1. 技术路线
研究以单只无指盘臭蛙(Odorrana grahami)皮肤为材料,共发现107条推定ESPs(16条获质谱验证),其中74条为该物种首次报道,含62条新肽序列(包括5条“孤儿肽”)。这些ESPs分属14个已知蛋白家族,其中11个家族为无指盘臭蛙首次报道,8个家族为臭蛙属首次发现。其中,蛙皮活性肽家族(FSAP,frog skin active peptide family)为多样性最丰富的家族(83条,含48条新序列),对比文献数据显示同一居群个体间存在高度多样性(图2)。至此,无指盘臭蛙FSAP成员累计达226条,为两栖动物之最(云南臭蛙O. andersonii 205条次之)。
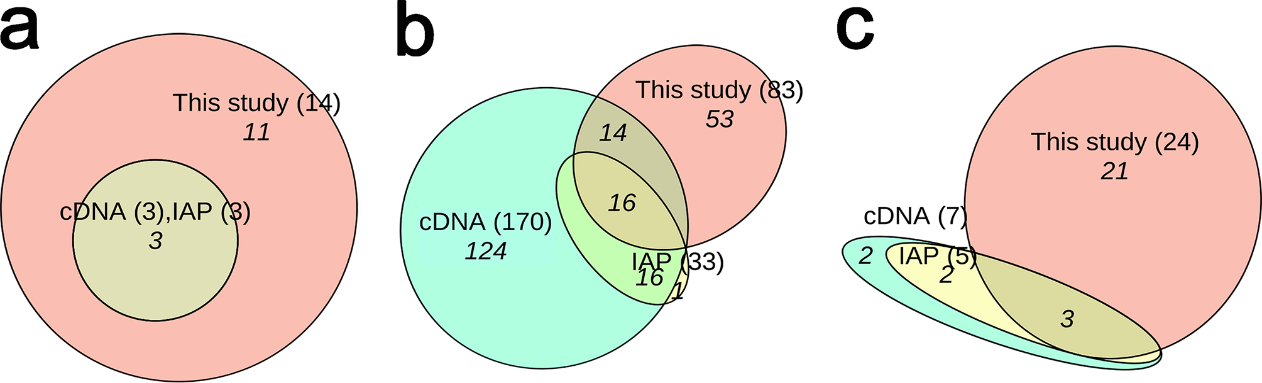
图2. 同一居群不同方法发现的无指盘臭蛙ESPs
(a) ESP家族分类 (b) FSAP家族成员 (c) 非FSAP家族成员。IAP指分离纯化法,cDNA指cDNA法
系统发育分析将FSAP成员划分为A-E五个进化枝,其中高变异的D枝包含多种非抗菌活性肽及两条与速激肽家族(tachykinin)趋同进化的肽(图3)。基于进化关系,本研究支持UniProt的蛋白家族分类标准,即将文献中激增的“无尾目特有抗菌肽家族”归并为少数家族(如FSAP、maximin-S、bombinin等)。
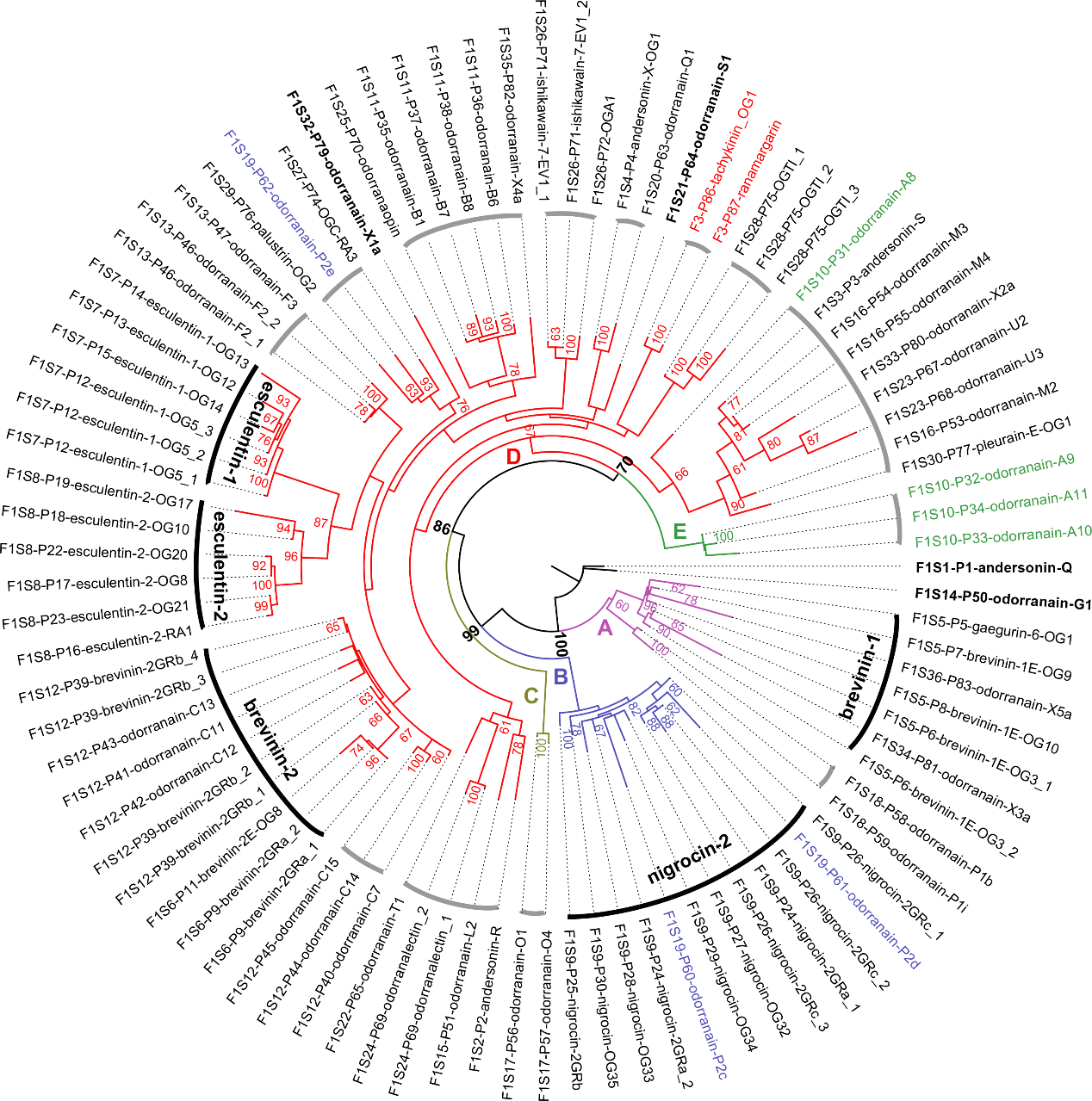
图3. FSAP 家族成员的系统发育树
该研究打破了臭蛙属皮肤ESPs研究长期局限于FSAP家族的格局,首次系统揭示其非FSAP家族肽的丰富多样性,证明即使在深入研究过的物种中仍有大量ESPs被遗漏,亟需在泛转录组学与泛肽组学框架下系统挖掘。该方法具有良好的可移植性,可广泛应用于其他真核生物,为两栖动物皮肤功能机制研究及新型药物先导化合物资源挖掘提供了重要技术平台和数据资源。
研究成果以“Integrative transcriptomics and peptidomics approach reveals unexpectedly diverse endogenous secretory peptides in Odorrana grahami frog skin”为题,近日在《BMC Biology》上发表。刘炯宇副研究员为第一作者兼共同通讯作者,江建平研究员为共同通讯作者。该研究获第二次青藏高原科考(STEP: 2019QZKK05010503)、中国生物多样性监测网络(Sino BON–两栖爬行动物)、四川省科技计划(2017JY0344)和国家自然科学基金(32200378)资助。
原文链接:https://link.springer.com/article/10.1186/s12915-025-02463-w




