中国微生物组数据平台发表整合重要项目数据
来源:《研究核酸》
作者:史文聿等
时间:2018-11-28
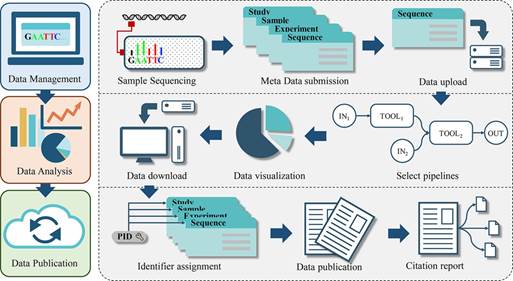
10月26日,《核酸研究》(Nucleic Acids Research)在线发表中国科学院微生物研究所微生物资源与大数据中心、世界微生物数据中心马俊才团队题为gcMeta: a Global Catalogue of Metagenomics platform to support the archiving, standardization and analysis of microbiome data的研究论文。gcMeta平台是一个微生物基因组及微生物组数据的管理、分析和发布平台,为国内外用户提供一站式的从数据存储、数据分析到数据发布的服务,目前已经整合了来自中科院微生物组计划及国内外多个重要项目的数据。该平台的发布将有效支撑我国微生物组研究并为未来我国国家微生物组计划的实施提供重要的支持。
近年来,美国、欧盟都陆续启动了微生物组相关的研究项目。但微生物组大数据的收集、存储、功能挖掘和开发利用一直是制约微生物组发展的核心问题。我国目前在微生物组数据管理中存在着标准不统一、缺乏跨领域的数据整合、高质量的参考数据库和数据的深度挖掘技术等问题。2017年,中科院启动了“中国微生物组计划”项目,项目旨在进一步强化中科院在微生物组研究和开发利用等方面的共性技术和平台优势,聚焦“人体健康和环境”微生物组,开发相应的微生物组学新方法、新技术;通过研究其结构与功能、群体间的竞争与合作,微生物组与人体等宿主和环境相互作用及与宿主的寄生共生健康发育等关系,发现微生物与人类和环境共同演化的科学规律。同时,也将在微生物组数据标准化的基础上,建立微生物组大数据计算、存储和共享平台,开发微生物组大数据挖掘的新方法,实现我国微生物组数据资源的系统管理和高效利用。
gcMeta建立了一个微生物基因组、元基因组和转录组管理、数据在线分析、可视化及数据发布的一站式系统。目前已经整合来自国际相关平台(NCBI、EBI、MG-RAST等)及重要项目(HMP、Tara等)超过12万样本数据,来自我国科学家的超过2000余个样本数据,总数据量超过120TB。平台为用户提供了多级的数据管理和权限控制体系,可用于各研究组管理未发表数据,并在研究组内共享,也可以将内部管理数据进行在线发布与公开。平台为所有公开数据提供基于Persistent Identifier (PID) (http://www.pidconsortium.eu/)系统的唯一PID号,用于在学术期刊的公开发表及后续数据引用及分析。此外,平台还整合了超过90个在线数据分析工具,提供针对扩增子序列、全基因组序列等4套分析工作流,所有的分析工具和工作流都是以web方式使用,方便微生物领域用户快速掌握及使用。用户可以通过该平台方便地实现数据管理、数据分析、结果展示和数据发布等一系列服务,平台也将为用户提供全过程的使用支持,欢迎国内外用户使用该平台。
微生物资源与大数据中心史文聿、亓合媛为论文共同第一作者,微生物资源与大数据中心及世界微生物数据中心马俊才及吴林寰为论文共同通讯作者。该研究得到中科院重点部署项目“人体与环境健康的微生物组共性技术研究”、国家重点研发计划“益生菌健康功能与基于肠道微生物组学的食品营养代谢机理研究”、中科院A类先导专项“地球大数据”、中科院信息化专项微生物领域云项目等的支持。(来源:中国科学院微生物研究所)
gcMeta: a Global Catalogue of Metagenomics platform to support the archiving, standardization and analysis of microbiome data
Abstract Meta-omics approaches have been increasingly used to study the structure and function of the microbial communities. A variety of large-scale collaborative projects are being conducted to encompass samples from diverse environments and habitats. This change has resulted in enormous demands for long-term data maintenance and capacity for data analysis. The Global Catalogue of Metagenomics (gcMeta) is a part of the ‘Chinese Academy of Sciences Initiative of Microbiome (CAS-CMI)’, which focuses on studying the human and environmental microbiome, establishing depositories of samples, strains and data, as well as promoting international collaboration. To accommodate and rationally organize massive datasets derived from several thousands of human and environmental microbiome samples, gcMeta features a database management system for archiving and publishing data in a standardized way. Another main feature is the integration of more than ninety web-based data analysis tools and workflows through a Docker platform which enables data analysis by using various operating systems. This platform has been rapidly expanding, and now hosts data from the CAS-CMI and a number of other ongoing research projects. In conclusion, this platform presents a powerful and user-friendly service to support worldwide collaborative efforts in the field of meta-omics research. This platform is freely accessible at https://gcmeta.wdcm.org/.
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gky1008/5144955?searchresult=1




