我国科技人员在杂合二倍体与跨物种基因组重排技术上取得重要进展
来源:《自然通讯》
作者:Michael J. Shen等
时间:2018-09-12
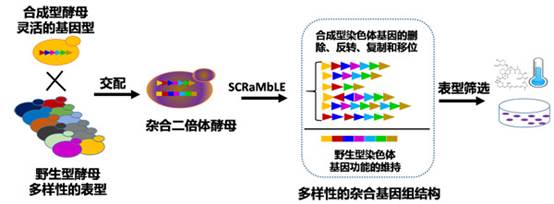
合成型基因组重排驱动酵母快速进化
遗传变异是生物进化的源泉,促使生物在亿万年间可以不断适应环境、不断进化。在生命科学领域,科学家开发出多种遗传变异技术,来获取多样的DNA,从而为获取多样的生物特征提供原料。然而以前的DNA变异技术大多只针对基因层面进行小规模改造,在更加复杂的基因组结构变异层面的人工构建技术仍具有挑战性。
最近,天津大学和纽约大学合作在Nature Communications杂志在线发表了题为“Heterozygous diploid and interspecies SCRaMbLEing”的研究论文。该研究首次将合成型酵母基因组(Sc2.0)的重排系统拓展到杂合二倍体和跨物种二倍体,并且利用基因组重排系统快速驱动细胞进化(模式图附后)。该技术一方面有助于加速工业微生物的性状改良,另一方面对于挖掘新的生物学知识有重要意义。
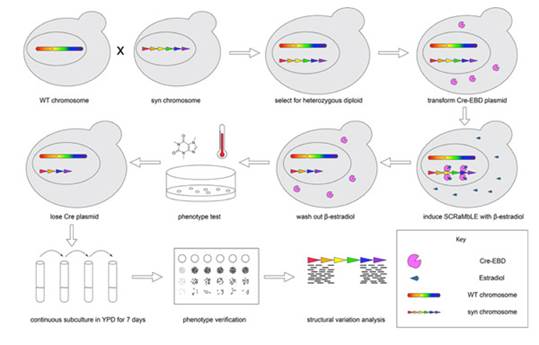
杂合二倍体与跨物种基因组重排模式图
本研究通过酵母交配的方式将具有灵活基因型的合成型酵母与具有多样化表型的野生型酵母相结合,使得SCRaMbLE(Synthetic Chromosome Recombination and Modification by LoxP-Mediated Evolution)系统驱动杂合二倍体和跨物种二倍体的基因组重排。研究人员将包含单条合成型染色体(synX)和包含两条合成型染色体(synV和synX)的单倍体酵母与来自酵母菌株库(SGRP)中的25株酿酒酵母(Saccharomyces cerevisiae)和27株奇艺酵母(Saccharomyces paradoxus)单倍体交配,获得100株菌的杂合二倍体和跨物种二倍体菌株库。

杂合二倍体和跨物种二倍体菌株库
科研人员使用一株酿清酒的酿酒酵母菌株Y12为例,通过杂合二倍体的基因组重排,研究人员成功获得两株在42度生长加快的重排菌株,全基因组测序分析其中一株重排菌包含5处大片段的删除和1处大片段的复制。研究结果表明,在二倍体中开启基因组重排系统比在单倍体中有更好的容忍度。研究人员又以一株奇艺酵母CBS5829为例,通过跨物种二倍体的基因组重排,成功获得十株咖啡因耐受性提升的重排菌株,全基因组测序分析独立的两株重排菌株中包含有相同片段的复制现象,验证实验显示复制片段中的POL32基因的加倍使酵母的咖啡因耐受性提升。
该技术一方面将有助于加速工业微生物的性状改良,另一方面将帮助挖掘新的基础生物学知识。该研究得到国家科技项目的支持。(来源:科技部 文中图片来源:天津大学元英进课题组)
Heterozygous diploid and interspecies SCRaMbLEing
Abstract SCRaMbLE (Synthetic Chromosome Rearrangement and Modification by LoxP-mediated Evolution) is a genome restructuring technique that can be used in synthetic genomes such as that of Sc2.0, the synthetic yeast genome, which contains hundreds to thousands of strategically positioned loxPsym sites. SCRaMbLE has been used to induce rearrangements in yeast strains harboring one or more synthetic chromosomes, as well as plasmid DNA in vitro and in vivo. Here we describe a collection of heterozygous diploid strains produced by mating haploid semisynthetic Sc2.0 strains to haploid native parental strains. We subsequently demonstrate that such heterozygous diploid strains are more robust to the effects of SCRaMbLE than haploid semisynthetic strains, rapidly improve rationally selected phenotypes in SCRaMbLEd heterozygous diploids, and establish that multiple sets of independent genomic rearrangements are able to lead to similar phenotype enhancements. Finally, we show that heterozygous diploid SCRaMbLE can also be carried out in interspecies hybrid strains.
原文链接:https://www.nature.com/articles/s41467-018-04157-0.pdf




