上海生科院利用 CRISPR/Cpf1 系统简单高效实现水稻多基因定点编辑
来源:Molecular Plant
作者:朱健康等
时间:2017-04-18
3 月 19 日,《分子植物》(Molecular Plant)杂志在线发表了中国科学院上海生命科学研究院植物逆境生物学研究中心朱健康研究组题为 Multiplex Gene Editing in Rice using the CRISPR-Cpf1 System 的研究论文。该工作在水稻中利用 CRISPR/Cpf1 系统实现多基因位点编辑,效率达到 40-75%;同时该系统在载体构建上比 CRISPR/Cas9 系统更加简单易行。该工作为水稻多基因定点编辑提供了一个简单高效的新工具。
CRISPR/Cas9 系统在基因定点编辑方面已经得到了大量的应用,然而 CRISPR/Cas9 系统依然存在着一定的局限性。在 CRISPR/Cas9 系统中,需要 CRISPR 转录形成 RNACRISPR RNA (CrRNA) 与反式作用型 CrRNA(trans-activating crRNA, tracrRNA) 两种小 RNA 分子,两种 RNA 分子部分区域配对形成复合体。随后,该复合体引导具有非特异核酸酶活性的 Cas 蛋白切割与 crRNA 匹配的 DNA 序列。因此,工程化的 CRISPR/Cas9 系统也需要将 CrRNA 与 tracrRNA 融合在一起形成单一的 chimeric RNA(chiRNA),并且需要在宿主 RNA 酶的帮助下才能发挥作用。同时,利用该系统进行单次多基因定点编辑时,载体系统构建复杂,每一个定点编辑位点均需要单独的启动子和终止系列。
近年来,科学家发现并改造出 Cpf1 (CRISPR from Prevotella and Francisella 1) 系统,研究表明该系统能克服 CRISPR/Cas9 的上述局限,它不需要 tracrRNA 的辅助,并且 Cpf1 蛋白兼具 DNA 剪切酶和 RNA 修剪酶的功能,不但能靶向切割 DNA 双链,而且能把相应的非成熟型 CrRNA(pre-crRNA) 加工剪切成成熟型 CrRNA。
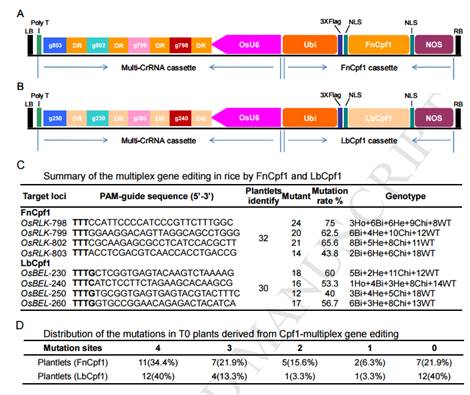
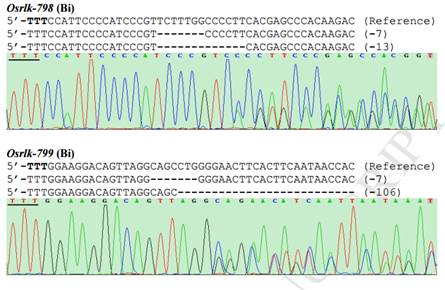
朱健康研究组利用 Francisella novicida Cpf1 (FnCpf1) 和 Lachnospiraceae bacterium ND2006 Cpf1 (LbCpf1) 对水稻进行单位点和多位点基因敲除的测试,研究表明上述两个 Cpf1 只需一条非常短的 20-21bp 的直接重复序列(direct repeats, DR)加上 22-24bp 的靶位点识别序列(guide)即可实现单基因敲除,更重要的是,把多个 DR-guide 单元直接串联,只需要一个启动子驱动即可简单高效地实现多基因敲除。该研究利用 4 个 DR-guide 单元组成的 CrRNA 短阵列分别对水稻 RLK 和 CYP81A 家族的四个基因进行编辑,各位点的敲除效率达到 40-75%。该系统简单、高效地在水稻中实现了多基因定点编辑,拓展了 CRISPR 系统在植物中的应用,为水稻基因组定点编辑提供了一个新利器。
该工作得到了中科院的经费支持。(来源:生物360)
Multiplex Gene Editing in Rice using the CRISPR-Cpf1 System
Abstract To compare the gene editing efficiency of Francisella novicida Cpf1 (FnCpf1) and Lachnospiraceae bacterium ND2006 Cpf1 (LbCpf1) in rice, two T-DNA constructs were designed based on the pCambia binary vector to express FnCpf1 and LbCpf1 systems (Supplemental Figure 1). We chose 22-24 nt target sequences to induce mutations at six sites from three endogenous genes: 5-Enolpyruvylshikimate 3-Phosphate Synthase (OsEPSPS, LOC_Os06g04280), Bentazon Sensitive Lethal (OsBEL, LOC_Os03g55240) and Phytoene Desaturase (OsPDS, LOC_Os03g08570). Our results showed that both FnCpf1 and LbCpf1 with their own mature DRs can introduce targeted gene mutations in transgenic plants (Supplemental Table 1). Mutations in OsPDS caused an albino phenotype, as expected (Supplemental Figure 2). Moreover, we found that the LbCpf1 system exhibited a higher editing efficiency than FnCpf1 in all of the six tested target sites, and both of them show big differences in the frequency of induced mutations between two target sites within the same gene (Supplemental Table 1)。
原文链接:http://www.sciencedirect.com/science/article/pii/S1674205217300710




